摘要:研究通过snm3C-seq结合snRNA-seq,首次绘制了人类皮下脂肪组织(SAT)的细胞类型分辨率表观基因组图谱。
多组学解析人类皮下脂肪细胞异质性
研究团队采用单核甲基化-3C测序(snm3C-seq)技术,对5例芬兰女性皮下脂肪组织(SAT)活检样本进行多组学分析,同步捕获6,652个核的DNA甲基化(mCG)和染色质构象数据,并与29,423个核的snRNA-seq数据整合。通过5kb分辨率mCG分析和100kb染色质互作聚类,鉴定出脂肪细胞、脂肪干细胞祖细胞(ASPCs)、血管周围细胞等7种主要细胞类型,并发现63个核呈现表观遗传"过渡态"特征——其染色质构象类似脂肪细胞而甲基化模式接近血管周围细胞,暗示人类脂肪细胞可能通过该路径分化。

图1 人类皮下脂肪组织的单细胞 DNA 甲基化组与三维基因组图谱
甲基化与转录组的跨模态调控网络
基因体mCG低甲基化与高表达呈现强相关性(如脂肪标记基因GPAM)。跨模态分析发现PPAR信号通路关键基因(ACSL1、ADIPOQ等)在脂肪细胞中同步呈现低mCG和高表达,而脂肪分化基因(如LEP、SREBF1)则富集于脂肪细胞特异性低甲基化区域。值得注意的是,仅21%的脂肪细胞标记基因在两种模态中重叠,但63%相关生物学通路(如脂质代谢)具有跨模态一致性,提示表观遗传与转录调控的协同作用。
髓系与脂肪细胞的甲基化"跷跷板"效应
全基因组共鉴定705,063个差异甲基化区域(DMRs),覆盖5.39%基因组。脂肪细胞与ASPCs分别呈现56.3%和50.6%区域低甲基化,而髓系细胞则73.0%区域高甲基化,47.3% DMRs呈现此消彼长模式。HOMER分析揭示髓系细胞低甲基化区域富集IRF4、MEF2B等转录因子结合基序,ENCODE的ChIP-seq数据验证这些基序在GM12878细胞中富集度达3.5倍(P<1×10-12)。
脂肪细胞特异的基因组三维结构
脂肪细胞表现出独特的染色质空间特征:短程互作(100kb-2Mb)比例较其他细胞高1.51倍(P<10-91),25kb分辨率下检测到更多结构域(平均4,120个/细胞)。染色体区室分析显示,脂肪细胞的A区室(活性染色质)与ADIPOQ等肥胖GWAS变异显著共定位,而髓系细胞A区室则富集炎症相关变异。值得注意的是,11,571个差异区室区域中,ADIPOQ基因所在基因组区域在ASPCs中处于B区室(抑制状态),而在脂肪细胞中转为A区室。
TET1-DNMT3A的表观遗传调控轴
单细胞转录组显示去甲基化酶TET1在脂肪细胞特异性高表达(P<10-300),而甲基转移酶DNMT3A在髓系细胞优势表达(P<10-136)。人类前脂肪细胞分化实验证实,TET1与PPARγ等脂肪生成基因呈现同步表达波动。SAT批量RNA-seq进一步发现,TET1表达与胰岛素敏感性正相关(Matsuda指数r=0.32),而DNMT3A则呈负相关(r=-0.28)。
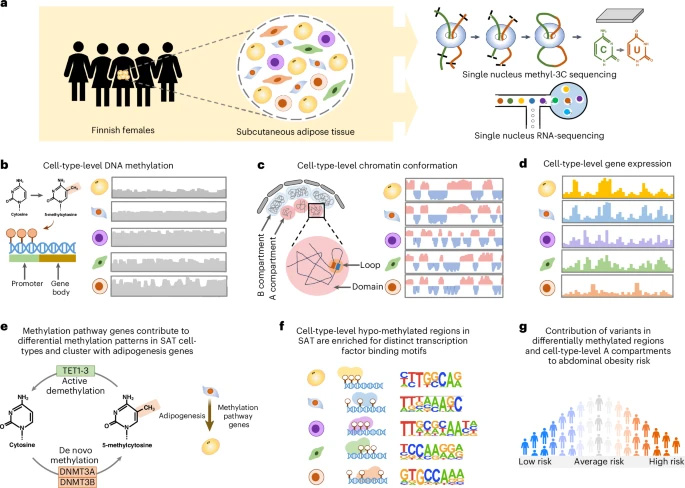
图2 本研究采用 snm3C-seq 和 snRNA-seq 技术,在人类皮下脂肪组织(SAT)中对细胞类型水平的 DNA 甲基化、染色质构象及基因表达进行表征,并解析腹部肥胖的遗传风险
肥胖风险的细胞类型特异性遗传基础
英国生物银行(UKB)39万人数据分析显示,脂肪细胞低甲基化区域和A区室的变异分别解释腰臀比调整BMI(WHRadjBMI)遗传风险的82%和79%,显著优于随机背景(P<0.001)。髓系细胞a区室变异则与c反应蛋白(crp)水平显著相关,印证了肥胖相关炎症的髓系细胞起源假说。gwas变异富集分析发现,脂肪细胞dmrs中肥胖相关变异达3.2倍富集(p=6.5×10-8)。
这项研究首次在单细胞层面揭示SAT表观遗传异质性,建立DNA甲基化-三维基因组-转录组的调控网络,为理解肥胖及其并发症的细胞类型特异性机制提供了新视角。发现的过渡态细胞群体和TET1-DNMT3A调控轴,为靶向干预代谢疾病提供了潜在新靶点。
参考资料
[1] Single-cell DNA methylome and 3D genome atlas of human subcutaneous adipose tissue
摘要:研究通过snm3C-seq结合snRNA-seq,首次绘制了人类皮下脂肪组织(SAT)的细胞类型分辨率表观基因组图谱。
多组学解析人类皮下脂肪细胞异质性
研究团队采用单核甲基化-3C测序(snm3C-seq)技术,对5例芬兰女性皮下脂肪组织(SAT)活检样本进行多组学分析,同步捕获6,652个核的DNA甲基化(mCG)和染色质构象数据,并与29,423个核的snRNA-seq数据整合。通过5kb分辨率mCG分析和100kb染色质互作聚类,鉴定出脂肪细胞、脂肪干细胞祖细胞(ASPCs)、血管周围细胞等7种主要细胞类型,并发现63个核呈现表观遗传"过渡态"特征——其染色质构象类似脂肪细胞而甲基化模式接近血管周围细胞,暗示人类脂肪细胞可能通过该路径分化。

图1 人类皮下脂肪组织的单细胞 DNA 甲基化组与三维基因组图谱
甲基化与转录组的跨模态调控网络
基因体mCG低甲基化与高表达呈现强相关性(如脂肪标记基因GPAM)。跨模态分析发现PPAR信号通路关键基因(ACSL1、ADIPOQ等)在脂肪细胞中同步呈现低mCG和高表达,而脂肪分化基因(如LEP、SREBF1)则富集于脂肪细胞特异性低甲基化区域。值得注意的是,仅21%的脂肪细胞标记基因在两种模态中重叠,但63%相关生物学通路(如脂质代谢)具有跨模态一致性,提示表观遗传与转录调控的协同作用。
髓系与脂肪细胞的甲基化"跷跷板"效应
全基因组共鉴定705,063个差异甲基化区域(DMRs),覆盖5.39%基因组。脂肪细胞与ASPCs分别呈现56.3%和50.6%区域低甲基化,而髓系细胞则73.0%区域高甲基化,47.3% DMRs呈现此消彼长模式。HOMER分析揭示髓系细胞低甲基化区域富集IRF4、MEF2B等转录因子结合基序,ENCODE的ChIP-seq数据验证这些基序在GM12878细胞中富集度达3.5倍(P<1×10-12)。
脂肪细胞特异的基因组三维结构
脂肪细胞表现出独特的染色质空间特征:短程互作(100kb-2Mb)比例较其他细胞高1.51倍(P<10-91),25kb分辨率下检测到更多结构域(平均4,120个/细胞)。染色体区室分析显示,脂肪细胞的A区室(活性染色质)与ADIPOQ等肥胖GWAS变异显著共定位,而髓系细胞A区室则富集炎症相关变异。值得注意的是,11,571个差异区室区域中,ADIPOQ基因所在基因组区域在ASPCs中处于B区室(抑制状态),而在脂肪细胞中转为A区室。
TET1-DNMT3A的表观遗传调控轴
单细胞转录组显示去甲基化酶TET1在脂肪细胞特异性高表达(P<10-300),而甲基转移酶DNMT3A在髓系细胞优势表达(P<10-136)。人类前脂肪细胞分化实验证实,TET1与PPARγ等脂肪生成基因呈现同步表达波动。SAT批量RNA-seq进一步发现,TET1表达与胰岛素敏感性正相关(Matsuda指数r=0.32),而DNMT3A则呈负相关(r=-0.28)。

图2 本研究采用 snm3C-seq 和 snRNA-seq 技术,在人类皮下脂肪组织(SAT)中对细胞类型水平的 DNA 甲基化、染色质构象及基因表达进行表征,并解析腹部肥胖的遗传风险
肥胖风险的细胞类型特异性遗传基础
英国生物银行(UKB)39万人数据分析显示,脂肪细胞低甲基化区域和A区室的变异分别解释腰臀比调整BMI(WHRadjBMI)遗传风险的82%和79%,显著优于随机背景(P<0.001)。髓系细胞a区室变异则与c反应蛋白(crp)水平显著相关,印证了肥胖相关炎症的髓系细胞起源假说。gwas变异富集分析发现,脂肪细胞dmrs中肥胖相关变异达3.2倍富集(p=6.5×10-8)。
这项研究首次在单细胞层面揭示SAT表观遗传异质性,建立DNA甲基化-三维基因组-转录组的调控网络,为理解肥胖及其并发症的细胞类型特异性机制提供了新视角。发现的过渡态细胞群体和TET1-DNMT3A调控轴,为靶向干预代谢疾病提供了潜在新靶点。
参考资料
[1] Single-cell DNA methylome and 3D genome atlas of human subcutaneous adipose tissue



