摘要:波兰弗罗茨瓦夫大学团队利用链霉菌模型开展SMC对ParB动态行为与功能调控的主题研究。
在细菌细胞中,染色体分离是保证遗传信息准确传递的核心生物学过程。这一过程主要由ParABS系统介导,其中ParB蛋白作为关键组分,通过结合特异的parS位点形成大型核蛋白复合体(称为segrosome),进而招募结构维持染色体(Structural Maintenance of Chromosomes, SMC)蛋白复合物来协调染色体的高级组织与分离。近年来研究发现ParB具有CTP酶活性,其通过CTP结合与水解循环调控自身与DNA的相互作用——CTP结合的ParB二聚体在parS位点成核后发生构象变化形成闭合钳结构,并沿DNA滑动实现扩散,而CTP水解则促使ParB从DNA解离。然而,SMC复合体作为ParB的重要招募对象,是否以及如何反馈调节ParB复合体的动态特性与功能,长期以来仍不明确。
针对该问题,弗罗茨瓦夫大学的Katarzyna Pawlikiewicz等研究人员以模式生物委内瑞拉链霉菌(Streptomyces venezuelae)为对象,通过构建ParB-HaloTag(ParB-HT)融合蛋白菌株,结合时间推移荧光显微镜、单分子追踪(SMT)、荧光恢复后漂白(FRAP)及染色质免疫沉淀测序(ChIP-seq)等多学科技术,系统探究了SMC存在与否对ParB动态行为、DNA结合特性及酶活性的影响。该研究论文已于2025年发表在《Nature Communications》期刊。

图1 SMC调控链霉菌中分离复合体内ParB的结合
研究主要依托以下关键技术方法:首先利用微生物遗传学手段构建了ParB-HT标签菌株及SMC敲除株(△smc),通过时间序列显微成像分析孢子形成过程中ParB复合体的动态变化;应用单分子追踪分析ParB蛋白的扩散运动;采用ChIP-seq在全基因组范围检测ParB与DNA的结合模式;并通过体外CTP水解实验量化SMC对ParB酶活性的调控。所有实验均设置野生型与突变体对照,样本来源于同步培养的链霉菌液体培养物。
研究结果主要包括以下方面:
The dynamics of ParB complexes in sporogenic cells
通过时间序列成像观察发现,在孢子形成细胞中,ParB-HT复合体在菌丝生长停止后27±10分钟(T1)开始呈现规则排列,随后在80分钟左右逐渐解离。该过程与细胞分裂蛋白FtsZ形成的Z环出现与消失时间高度同步,表明ParB复合体在孢子形成期中短暂存在,并于细胞分裂时发生解离。
Elimination of SMC promotes disassembly of ParB-HT complexes in sporogenic cells
在SMC缺失菌株(△smc)中,ParB复合体规则排列的出现时间略早于野生型(20±9分钟),但其解离时间显著提前(79±25分钟 vs 野生型114±42分钟),且存在时间变异性降低。此外,SMC缺失还导致隔膜形成延迟和孢子成型时间略微延长。Western blotting排除了ParB蛋白水平变化的影响,表明SMC确实具有稳定ParB复合体的作用。
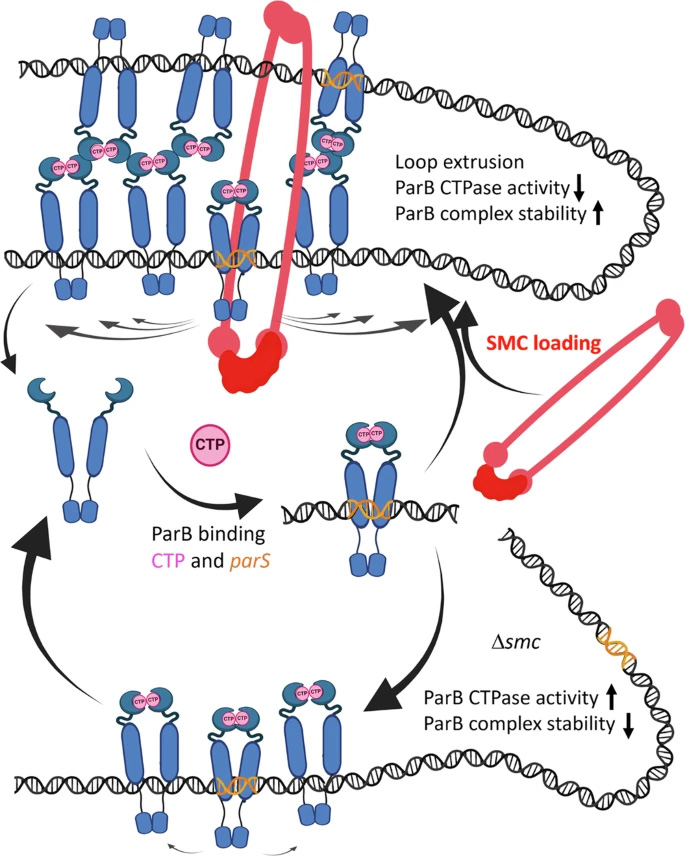
图2 SMC的加载对ParB复合体组装产生正反馈调节
Elimination of SMC lowers mobility of ParB molecules
单分子追踪显示,在孢子形成早期(17小时),SMC缺失并未改变ParB的移动性;但在晚期(22小时),△smc菌株中ParB的平均扩散系数显著降低(0.045μm2/s vs 野生型0.106μm2/s),且固定分子比例升高(62% vs 41.5%)。在幼龄营养菌丝中,SMC缺失同样导致ParB移动性下降和固定分子分布分散化,提示SMC缺失可能增强了ParB与非特异性DNA的结合。
The absence of SMC increases the turnover of ParB-HT in the apical complex FRAP实验表明,尽管SMC缺失菌株与野生型均呈现约60%的荧光恢复率,但前者恢复半衰期(tau)显著缩短(135±72秒 vs 222±200秒),说明SMC缺失环境下ParB复合体具有更快的周转速率,与前述复合体稳定性下降的结果一致。
SMC promotes ParB spreading at parS sites
ChIP-seq分析揭示,在野生型中ParB结合于16个已知parS位点及1个新位点,并在其上下游延伸2.5–3 kb,表现出典型的扩散特征。而在△smc菌株中,ParB在parS位点的结合信号减弱至野生型的一半,扩散现象几乎消失,但全染色体范围内的非特异性结合反而增加。这表明SMC是ParB特异性结合parS并实现扩散的关键促进因子。
SMC reduces ParB CTPase activity
体外酶活实验表明,在parS质粒存在下,SMC的添加使ParB的CTP水解活性从65.1±2.9 μmol/μmol/h降至35.2±5.2 μmol/μmol/h。利用蛋白互作界面突变体ParBSMC-(TDR132-134→AAT及D164R)进一步验证发现,该突变体本身酶活性降低,且SMC对其活性的抑制作用显著减弱,说明SMC对ParB CTP酶活的抑制依赖于二者间的直接相互作用。
研究最终得出结论:SMC通过直接相互作用抑制ParB的CTP水解活性,从而稳定其闭合钳构象,促进ParB在parS位点的加载与沿DNA的扩散,增强segrosome的稳定性。这一正反馈调控机制对于孢子形成期染色体的高效分离具有关键作用。同时,该研究揭示了SMC-ParB互作在细菌染色体组织中的核心地位,为理解ParABS系统与SMC协同工作机制提供了重要理论依据。
参考资料
[1] SMC modulates ParB engagement in segregation complexes in streptomyces
摘要:波兰弗罗茨瓦夫大学团队利用链霉菌模型开展SMC对ParB动态行为与功能调控的主题研究。
在细菌细胞中,染色体分离是保证遗传信息准确传递的核心生物学过程。这一过程主要由ParABS系统介导,其中ParB蛋白作为关键组分,通过结合特异的parS位点形成大型核蛋白复合体(称为segrosome),进而招募结构维持染色体(Structural Maintenance of Chromosomes, SMC)蛋白复合物来协调染色体的高级组织与分离。近年来研究发现ParB具有CTP酶活性,其通过CTP结合与水解循环调控自身与DNA的相互作用——CTP结合的ParB二聚体在parS位点成核后发生构象变化形成闭合钳结构,并沿DNA滑动实现扩散,而CTP水解则促使ParB从DNA解离。然而,SMC复合体作为ParB的重要招募对象,是否以及如何反馈调节ParB复合体的动态特性与功能,长期以来仍不明确。
针对该问题,弗罗茨瓦夫大学的Katarzyna Pawlikiewicz等研究人员以模式生物委内瑞拉链霉菌(Streptomyces venezuelae)为对象,通过构建ParB-HaloTag(ParB-HT)融合蛋白菌株,结合时间推移荧光显微镜、单分子追踪(SMT)、荧光恢复后漂白(FRAP)及染色质免疫沉淀测序(ChIP-seq)等多学科技术,系统探究了SMC存在与否对ParB动态行为、DNA结合特性及酶活性的影响。该研究论文已于2025年发表在《Nature Communications》期刊。

图1 SMC调控链霉菌中分离复合体内ParB的结合
研究主要依托以下关键技术方法:首先利用微生物遗传学手段构建了ParB-HT标签菌株及SMC敲除株(△smc),通过时间序列显微成像分析孢子形成过程中ParB复合体的动态变化;应用单分子追踪分析ParB蛋白的扩散运动;采用ChIP-seq在全基因组范围检测ParB与DNA的结合模式;并通过体外CTP水解实验量化SMC对ParB酶活性的调控。所有实验均设置野生型与突变体对照,样本来源于同步培养的链霉菌液体培养物。
研究结果主要包括以下方面:
The dynamics of ParB complexes in sporogenic cells
通过时间序列成像观察发现,在孢子形成细胞中,ParB-HT复合体在菌丝生长停止后27±10分钟(T1)开始呈现规则排列,随后在80分钟左右逐渐解离。该过程与细胞分裂蛋白FtsZ形成的Z环出现与消失时间高度同步,表明ParB复合体在孢子形成期中短暂存在,并于细胞分裂时发生解离。
Elimination of SMC promotes disassembly of ParB-HT complexes in sporogenic cells
在SMC缺失菌株(△smc)中,ParB复合体规则排列的出现时间略早于野生型(20±9分钟),但其解离时间显著提前(79±25分钟 vs 野生型114±42分钟),且存在时间变异性降低。此外,SMC缺失还导致隔膜形成延迟和孢子成型时间略微延长。Western blotting排除了ParB蛋白水平变化的影响,表明SMC确实具有稳定ParB复合体的作用。

图2 SMC的加载对ParB复合体组装产生正反馈调节
Elimination of SMC lowers mobility of ParB molecules
单分子追踪显示,在孢子形成早期(17小时),SMC缺失并未改变ParB的移动性;但在晚期(22小时),△smc菌株中ParB的平均扩散系数显著降低(0.045μm2/s vs 野生型0.106μm2/s),且固定分子比例升高(62% vs 41.5%)。在幼龄营养菌丝中,SMC缺失同样导致ParB移动性下降和固定分子分布分散化,提示SMC缺失可能增强了ParB与非特异性DNA的结合。
The absence of SMC increases the turnover of ParB-HT in the apical complex FRAP实验表明,尽管SMC缺失菌株与野生型均呈现约60%的荧光恢复率,但前者恢复半衰期(tau)显著缩短(135±72秒 vs 222±200秒),说明SMC缺失环境下ParB复合体具有更快的周转速率,与前述复合体稳定性下降的结果一致。
SMC promotes ParB spreading at parS sites
ChIP-seq分析揭示,在野生型中ParB结合于16个已知parS位点及1个新位点,并在其上下游延伸2.5–3 kb,表现出典型的扩散特征。而在△smc菌株中,ParB在parS位点的结合信号减弱至野生型的一半,扩散现象几乎消失,但全染色体范围内的非特异性结合反而增加。这表明SMC是ParB特异性结合parS并实现扩散的关键促进因子。
SMC reduces ParB CTPase activity
体外酶活实验表明,在parS质粒存在下,SMC的添加使ParB的CTP水解活性从65.1±2.9 μmol/μmol/h降至35.2±5.2 μmol/μmol/h。利用蛋白互作界面突变体ParBSMC-(TDR132-134→AAT及D164R)进一步验证发现,该突变体本身酶活性降低,且SMC对其活性的抑制作用显著减弱,说明SMC对ParB CTP酶活的抑制依赖于二者间的直接相互作用。
研究最终得出结论:SMC通过直接相互作用抑制ParB的CTP水解活性,从而稳定其闭合钳构象,促进ParB在parS位点的加载与沿DNA的扩散,增强segrosome的稳定性。这一正反馈调控机制对于孢子形成期染色体的高效分离具有关键作用。同时,该研究揭示了SMC-ParB互作在细菌染色体组织中的核心地位,为理解ParABS系统与SMC协同工作机制提供了重要理论依据。
参考资料
[1] SMC modulates ParB engagement in segregation complexes in streptomyces



