摘要:每一个CRISPR反应的核心,都是通过引导RNA到DNA上的目标位点的Cas蛋白质的强分子连接。
每一个CRISPR反应的核心,无论是自然发生在细菌中,还是通过CRIPSR-Cas基因编辑技术加以利用,都是通过引导RNA到达DNA上的目标位点,使Cas蛋白质形成一个强大的分子键。
霍华德·休斯医学研究所研究员Michelle Wang说:“在稳定的边界和在正确的时间脱离之间有一个平衡。”“我们真正想要的是调节结合力的能力。这给了我们微调基因编辑潜力的可能性。”
Wang实验室的博士生、该论文的主要作者Porter Hall认为,Cas蛋白的结合不能太短暂。如果它不能稳定地结合DNA的目标区域,精确的基因编辑可能不会有效,可能会导致脱靶效应。“但如果蛋白质永远停留在那里,那么基因编辑过程就无法完成,”Hall说。

图1 RNA聚合酶如何可以不进行切割就识别DNA序列就去除一个结合的dCas 图源:[1])
通过对Cas与DNA结合的精确分子水平机制的研究,Wang和他的同事们首次解释了运动蛋白(RNA聚合酶)如何去除一个结合的dCas(一种经过工程改造的Cas),可以不进行切割就识别DNA序列。
这一见解揭示了如何调整Cas去除,有助于未来的CRISPR应用。
研究人员写道:“要充分发挥CRISPR技术的潜力,对Cas结合稳定性的深入机制理解至关重要。”“这项工作强调了R-loop在dCas结合稳定性中的重要性,并为CRISPR技术的广泛应用提供了有价值的机制见解。”
Wang实验室研究马达蛋白如何沿着DNA链移动,执行重要的生物过程。
Wang说,马达蛋白RNA聚合酶在执行基因表达功能时,会对“路障”施加压力,将DNA复制到RNA。在本研究中,这个路障就是内切酶缺陷Cas (dCas)。
此前,研究人员使用纳米光子镊子机械地分离了两条DNA链,绘制出结合的dCas蛋白质在DNA上的位置。他们称之为DNA unzipping mapper。
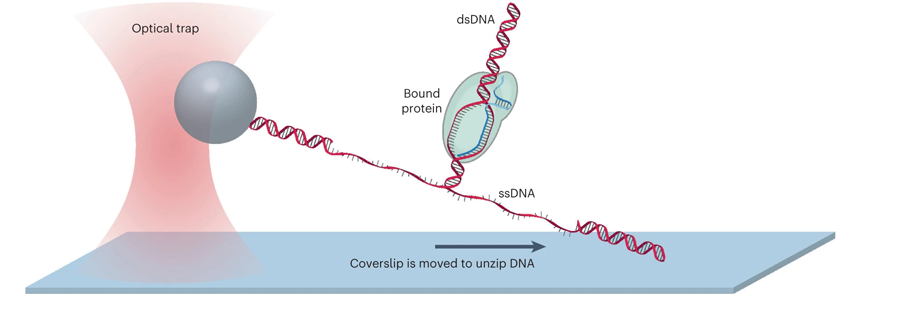
图2 使用DNA解压缩映射器绘制dCas与DNA相互作用的高分辨率图 (图源:[1])
先前的研究表明,马达蛋白只可能从一侧(极性)去除dCas。使用当前研究的DNA unzipping mapper,康奈尔大学的研究人员发现了其中的原因:因为RNA聚合酶只能从一侧折叠结合dCas的引导RNA和目标DNA之间形成的环(称为 "R-loop"),即距离PAM(protospacer adjacent motif)远的一侧(或远处),PAM是一个2-6个碱基对长的短DNA序列,紧随裂解目标DNA区域。
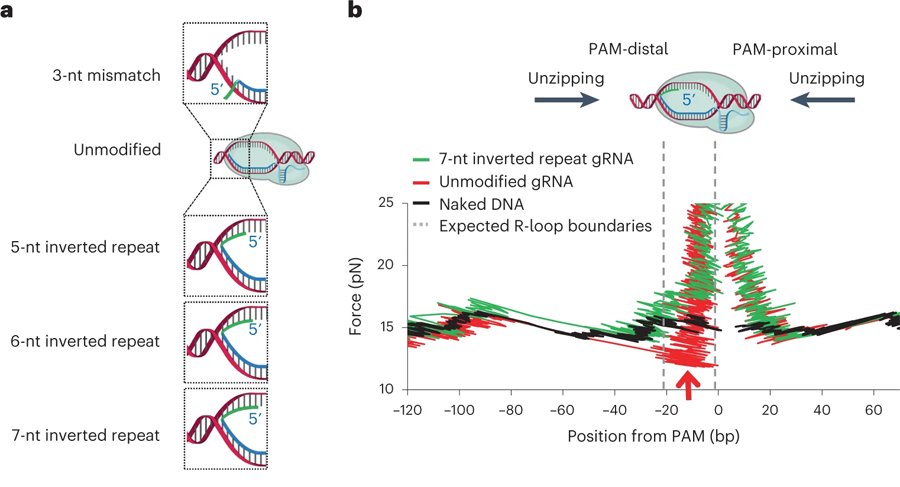
图3 结合dCas复合物的转录读取的定量测定(图源:[1])
研究人员描述了该机制是如何工作的,他们也展示了如何通过修改引导RNA来调整dCas R-loop的稳定性。
“我们希望Cas蛋白质如何工作的基础知识最终能导致更有效的基因编辑和更广泛的CRISPR技术应用,”Wang说。
参考资料:
[1] Polarity of the CRISPR Roadblock to Transcription
摘要:每一个CRISPR反应的核心,都是通过引导RNA到DNA上的目标位点的Cas蛋白质的强分子连接。
每一个CRISPR反应的核心,无论是自然发生在细菌中,还是通过CRIPSR-Cas基因编辑技术加以利用,都是通过引导RNA到达DNA上的目标位点,使Cas蛋白质形成一个强大的分子键。
霍华德·休斯医学研究所研究员Michelle Wang说:“在稳定的边界和在正确的时间脱离之间有一个平衡。”“我们真正想要的是调节结合力的能力。这给了我们微调基因编辑潜力的可能性。”
Wang实验室的博士生、该论文的主要作者Porter Hall认为,Cas蛋白的结合不能太短暂。如果它不能稳定地结合DNA的目标区域,精确的基因编辑可能不会有效,可能会导致脱靶效应。“但如果蛋白质永远停留在那里,那么基因编辑过程就无法完成,”Hall说。

图1 RNA聚合酶如何可以不进行切割就识别DNA序列就去除一个结合的dCas 图源:[1])
通过对Cas与DNA结合的精确分子水平机制的研究,Wang和他的同事们首次解释了运动蛋白(RNA聚合酶)如何去除一个结合的dCas(一种经过工程改造的Cas),可以不进行切割就识别DNA序列。
这一见解揭示了如何调整Cas去除,有助于未来的CRISPR应用。
研究人员写道:“要充分发挥CRISPR技术的潜力,对Cas结合稳定性的深入机制理解至关重要。”“这项工作强调了R-loop在dCas结合稳定性中的重要性,并为CRISPR技术的广泛应用提供了有价值的机制见解。”
Wang实验室研究马达蛋白如何沿着DNA链移动,执行重要的生物过程。
Wang说,马达蛋白RNA聚合酶在执行基因表达功能时,会对“路障”施加压力,将DNA复制到RNA。在本研究中,这个路障就是内切酶缺陷Cas (dCas)。
此前,研究人员使用纳米光子镊子机械地分离了两条DNA链,绘制出结合的dCas蛋白质在DNA上的位置。他们称之为DNA unzipping mapper。

图2 使用DNA解压缩映射器绘制dCas与DNA相互作用的高分辨率图 (图源:[1])
先前的研究表明,马达蛋白只可能从一侧(极性)去除dCas。使用当前研究的DNA unzipping mapper,康奈尔大学的研究人员发现了其中的原因:因为RNA聚合酶只能从一侧折叠结合dCas的引导RNA和目标DNA之间形成的环(称为 "R-loop"),即距离PAM(protospacer adjacent motif)远的一侧(或远处),PAM是一个2-6个碱基对长的短DNA序列,紧随裂解目标DNA区域。

图3 结合dCas复合物的转录读取的定量测定(图源:[1])
研究人员描述了该机制是如何工作的,他们也展示了如何通过修改引导RNA来调整dCas R-loop的稳定性。
“我们希望Cas蛋白质如何工作的基础知识最终能导致更有效的基因编辑和更广泛的CRISPR技术应用,”Wang说。
参考资料:
[1] Polarity of the CRISPR Roadblock to Transcription



