摘要:加州大学圣地亚哥分校的研究人员利用单细胞RNA测序发现了一种“分子指纹”,可以预测神经元在损伤后是否会再生,这为理解和增强神经元再生提供了新的见解。
神经元是构成我们大脑和脊髓的主要细胞,它是受伤后再生最慢的细胞之一,许多神经元不能完全再生。虽然科学家们在理解神经元再生方面取得了进展,但仍不清楚为什么有些神经元可以再生,而有些则不能。
加州大学圣地亚哥分校医学院的研究人员利用单细胞RNA测序(一种确定单个细胞中哪些基因被激活的方法),发现了一种新的生物标志物,可用于预测神经元在损伤后是否会再生。他们在小鼠身上测试了他们的发现,发现这种生物标志物在整个神经系统和不同发育阶段的神经元中始终是可靠的。这项研究于2023年10月16日发表在《Neuron》杂志上。

图1 深层scRNA测序揭示了一种广泛应用的再生分类器
这篇文章主要介绍了通过深度单细胞RNA测序技术,研究脊髓损伤后皮质脊髓束(CST)神经元的再生能力。通过对再生的CST神经元进行高通量测序,研究人员建立了一个名为“再生分类器”(Regeneration Classifier)的预测模型,能够预测不同神经元类型的再生潜力。该模型可以应用于不同发育阶段或损伤后的神经元,为研究神经元再生提供了一种新的工具。
此外,文章还通过网络分析,发现抗氧化反应和线粒体生物发生是再生过程中重要的生物学过程。进一步的研究证实,抗氧化反应基因NFE2L2(或NRF2)在CST再生中起着关键作用。这些发现揭示了抗氧化反应在神经元再生中的重要性,并为未来的治疗提供了潜在的靶点。
单细胞测序的力量
加州大学圣地亚哥分校医学院神经科学系教授、资深作者Binhai Zheng博士说:“单细胞测序技术正在帮助我们比以往任何时候都更详细地研究神经元的生物学,这项研究确实证明了这种能力。我们在这里发现的可能只是基于单细胞数据的新一代复杂生物标志物的开始。”
研究人员将重点放在皮质脊髓束的神经元上,皮质脊髓束是中枢神经系统中帮助控制运动的关键部分。损伤后,这些神经元是最不可能再生轴突的,轴突是神经元用来相互交流的细长结构。这就是为什么大脑和脊髓的损伤是毁灭性的。“如果你的手臂或腿受伤了,这些神经可以再生,而且通常有可能完全恢复功能,但中枢神经系统却不是这样,”第一作者Hugo Kim博士说。“从大多数大脑和脊髓损伤中恢复非常困难,因为这些细胞的再生能力非常有限。一旦他们走了,他们就走了。”
识别生物标志物
研究人员使用单细胞RNA测序来分析脊髓损伤小鼠神经元中的基因表达。他们使用现有的分子技术鼓励这些神经元再生,但最终,这只对一部分细胞起作用。这种实验设置允许研究人员比较再生和非再生神经元的测序数据。
此外,通过关注相对较少数量的细胞——只有300多个——研究人员能够非常仔细地观察每个细胞。“就像每个人都不一样,每个细胞都有自己独特的生物学特性,”Zheng说。“探索细胞之间的细微差异可以告诉我们很多关于这些细胞如何工作的信息。”
利用计算机算法分析他们的测序数据,研究人员发现了一种独特的基因表达模式,可以预测单个神经元在受伤后最终是否会再生。这种模式还包括一些以前从未涉及到神经元再生的基因。
“这就像神经元再生的分子指纹,”Zheng补充说。
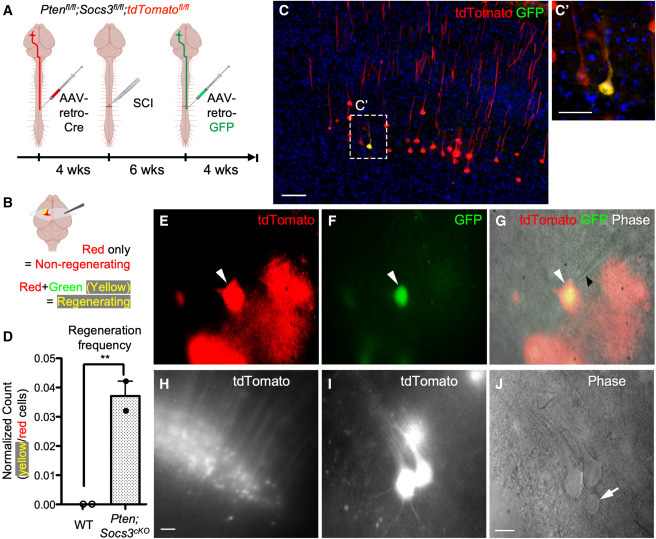
图2 通过深度单细胞RNA测序技术,研究脊髓损伤后皮质脊髓束(CST)神经元的再生能力
验证再生分类器
为了验证他们的发现,研究人员在26个已发表的单细胞RNA测序数据集上测试了这种分子指纹,他们将其命名为再生分类器。这些数据集包括来自神经系统不同部分和不同发育阶段的神经元。
研究小组发现,除了少数例外,再生分类器成功地预测了单个神经元的再生潜力,并能够重现以前研究中已知的趋势,比如出生后神经元再生急剧减少。
Zheng说:“通过对来自完全不同研究方向的许多组数据的验证,我们发现了神经元再生的潜在生物学基础。我们需要做更多的工作来完善我们的方法,但我认为我们已经发现了一种模式,可以适用于所有再生神经元。”
虽然在小鼠身上的结果很有希望,但研究人员警告说,目前,再生分类器是一个帮助实验室神经科学研究人员的工具,而不是临床患者的诊断测试。Zheng说:“在临床环境中使用单细胞测序仍然有很多障碍,比如成本高,分析大量数据困难,最重要的是,获取感兴趣的组织。目前,我们有兴趣探索如何在临床前环境中使用再生分类器来预测新的再生疗法的有效性,并帮助这些疗法更接近临床试验。”
参考资料
[1] Deep scRNA sequencing reveals a broadly applicable Regeneration Classifier and implicates antioxidant response in corticospinal axon regeneration
摘要:加州大学圣地亚哥分校的研究人员利用单细胞RNA测序发现了一种“分子指纹”,可以预测神经元在损伤后是否会再生,这为理解和增强神经元再生提供了新的见解。
神经元是构成我们大脑和脊髓的主要细胞,它是受伤后再生最慢的细胞之一,许多神经元不能完全再生。虽然科学家们在理解神经元再生方面取得了进展,但仍不清楚为什么有些神经元可以再生,而有些则不能。
加州大学圣地亚哥分校医学院的研究人员利用单细胞RNA测序(一种确定单个细胞中哪些基因被激活的方法),发现了一种新的生物标志物,可用于预测神经元在损伤后是否会再生。他们在小鼠身上测试了他们的发现,发现这种生物标志物在整个神经系统和不同发育阶段的神经元中始终是可靠的。这项研究于2023年10月16日发表在《Neuron》杂志上。

图1 深层scRNA测序揭示了一种广泛应用的再生分类器
这篇文章主要介绍了通过深度单细胞RNA测序技术,研究脊髓损伤后皮质脊髓束(CST)神经元的再生能力。通过对再生的CST神经元进行高通量测序,研究人员建立了一个名为“再生分类器”(Regeneration Classifier)的预测模型,能够预测不同神经元类型的再生潜力。该模型可以应用于不同发育阶段或损伤后的神经元,为研究神经元再生提供了一种新的工具。
此外,文章还通过网络分析,发现抗氧化反应和线粒体生物发生是再生过程中重要的生物学过程。进一步的研究证实,抗氧化反应基因NFE2L2(或NRF2)在CST再生中起着关键作用。这些发现揭示了抗氧化反应在神经元再生中的重要性,并为未来的治疗提供了潜在的靶点。
单细胞测序的力量
加州大学圣地亚哥分校医学院神经科学系教授、资深作者Binhai Zheng博士说:“单细胞测序技术正在帮助我们比以往任何时候都更详细地研究神经元的生物学,这项研究确实证明了这种能力。我们在这里发现的可能只是基于单细胞数据的新一代复杂生物标志物的开始。”
研究人员将重点放在皮质脊髓束的神经元上,皮质脊髓束是中枢神经系统中帮助控制运动的关键部分。损伤后,这些神经元是最不可能再生轴突的,轴突是神经元用来相互交流的细长结构。这就是为什么大脑和脊髓的损伤是毁灭性的。“如果你的手臂或腿受伤了,这些神经可以再生,而且通常有可能完全恢复功能,但中枢神经系统却不是这样,”第一作者Hugo Kim博士说。“从大多数大脑和脊髓损伤中恢复非常困难,因为这些细胞的再生能力非常有限。一旦他们走了,他们就走了。”
识别生物标志物
研究人员使用单细胞RNA测序来分析脊髓损伤小鼠神经元中的基因表达。他们使用现有的分子技术鼓励这些神经元再生,但最终,这只对一部分细胞起作用。这种实验设置允许研究人员比较再生和非再生神经元的测序数据。
此外,通过关注相对较少数量的细胞——只有300多个——研究人员能够非常仔细地观察每个细胞。“就像每个人都不一样,每个细胞都有自己独特的生物学特性,”Zheng说。“探索细胞之间的细微差异可以告诉我们很多关于这些细胞如何工作的信息。”
利用计算机算法分析他们的测序数据,研究人员发现了一种独特的基因表达模式,可以预测单个神经元在受伤后最终是否会再生。这种模式还包括一些以前从未涉及到神经元再生的基因。
“这就像神经元再生的分子指纹,”Zheng补充说。

图2 通过深度单细胞RNA测序技术,研究脊髓损伤后皮质脊髓束(CST)神经元的再生能力
验证再生分类器
为了验证他们的发现,研究人员在26个已发表的单细胞RNA测序数据集上测试了这种分子指纹,他们将其命名为再生分类器。这些数据集包括来自神经系统不同部分和不同发育阶段的神经元。
研究小组发现,除了少数例外,再生分类器成功地预测了单个神经元的再生潜力,并能够重现以前研究中已知的趋势,比如出生后神经元再生急剧减少。
Zheng说:“通过对来自完全不同研究方向的许多组数据的验证,我们发现了神经元再生的潜在生物学基础。我们需要做更多的工作来完善我们的方法,但我认为我们已经发现了一种模式,可以适用于所有再生神经元。”
虽然在小鼠身上的结果很有希望,但研究人员警告说,目前,再生分类器是一个帮助实验室神经科学研究人员的工具,而不是临床患者的诊断测试。Zheng说:“在临床环境中使用单细胞测序仍然有很多障碍,比如成本高,分析大量数据困难,最重要的是,获取感兴趣的组织。目前,我们有兴趣探索如何在临床前环境中使用再生分类器来预测新的再生疗法的有效性,并帮助这些疗法更接近临床试验。”
参考资料
[1] Deep scRNA sequencing reveals a broadly applicable Regeneration Classifier and implicates antioxidant response in corticospinal axon regeneration



